UNet++: Redesigning Skip Connections to ExploitMultiscale Features in Image Segmentation中文阅读
本文最后更新于:2023年4月7日 下午
阅读的第二篇论文,翻译的一般,英文好的,希望阅读原文。
UNet++: Redesigning Skip Connections to ExploitMultiscale Features in Image Segmentation
Zongwei Zhou, Member, IEEE, Md Mahfuzur Rahman Siddiquee, Member, IEEE, Nima Tajbakhsh, Member, IEEE, and Jianming Liang, Senior Member, IEEE
摘要:
医学图像分割的最新模型是U-Net和完全卷积网络(FCN)的变体。尽管取得了成功,但是这两个模型有两个局限性:(1)最佳深度是不知道的,需要宽的结构探索或者效率低的不同深度模型集成; (2)它们的skip connections施加了不必要的限制性融合方案,仅在编码器和解码器子网相同比例的特征图上强制聚合。为克服这两个限制,我们提出了UNet ++,这是一种新的神经网络架构,用于语义分割和实例分割。(1)有效的缓解了未知的网络深度,并且都是不同深度的U-Net的集合,这些U-Net共享一个编码器和同时使用深度监督进行合作学习; (2)重新设计skip connections以在解码器子网络上聚合不同尺度的语义特征,从而产生一个高度灵活的特征融合方案(3)设计一个剪枝方案以加快UNet ++的推理速度。我们使用六个不同的医学图像分割数据集对UNet ++进行了评估,涵盖了多种成像方式,例如计算机断层扫描(CT),磁共振成像(MRI),和电子显微镜(EM),并证明了(1)UNet ++对于交叉了不同数据集和主干架构的语义分割任务,其始终优于baseline模型,; (2)UNet ++提高了各种尺寸对象的分割质量,这是对固定深度U-Net的改进; (3)Mask RCNN ++(具有UNet ++设计的Mask R-CNN)在执行实例任务方面优于原始Mask R-CNN分割; (4)剪枝过的UNet ++模型实现了明显的加速,同时仅显示出适度的性能下降。可在https://github.com/MrGiovanni/UNetPlusPlus上获得我们的实施和预训练模型。
索引词:神经结构分割,肝脏分割,细胞分割,核分割,脑肿瘤分割,肺结节分割,医学图像分割,语义分割,实例分割,深度监督,模型修剪。
I. I NTRODUCTION
编码器-解码器网络广泛用于现代语义分割和实例分割模型[1],[2],[3],[4],[5],[6]。 它们的成功很大程度上归功于其skip connections,skip connections 将解码器子网中的深度,语义,粗粒度特征图与编码器子网中的浅,低层次,细粒度特征图结合在一起,并被证明在恢复目标对象[7],[8],[9]细粒度细节是有效的, 即使在复杂的情况下[10],[11]。 skip connections在实例级分割模型(例如[12],[13])的成功中也发挥了关键的作用,其中的想法是对所需对象的每个实例进行分割和区分。
然而,这些用于图像分割的编码器-解码器架构具有两个限制。首先,编码器/解码器网络的最佳深度可能因一个应用程序而异,取决于任务难度和可用于训练的标记数据量。一种简单的方法是分别训练不同深度的模型,然后在推理时间期间[14],[15],[16]将结果模型合在一起。但是,从部署的角度来看,这种简单的方法效率低下,因为这些网络并不共享公共的编码器。此外,由于独立训练,这些网络无法享受来自多任务学习的优势[17],[18]。其次,在编码器-解码器网络中使用的skip connections的设计受到不必要的限制,要求融合相同比例的编码器和解码器特征图。尽管是自然设计,但来自解码器和编码器网络相同比例的特征图在语义上是不同的,没有可靠的理论可以保证它们是特征融合的最佳匹配。
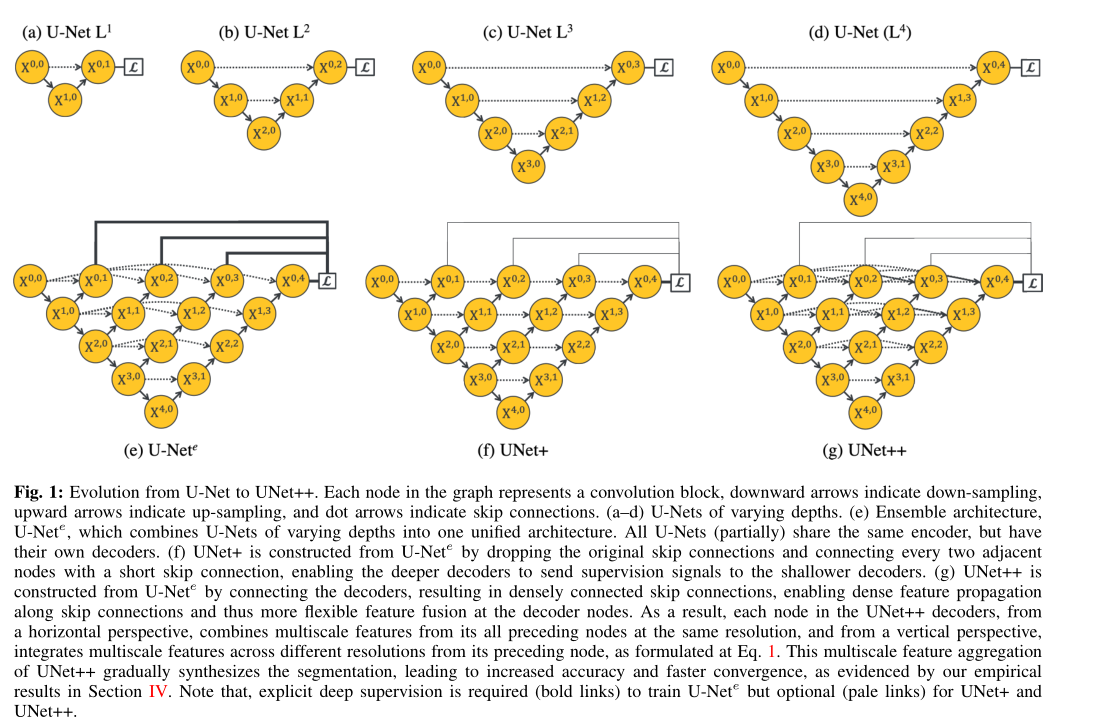
在本文中,我们介绍了UNet ++,这是一种旨在克服以上限制的新型通用图像分割体系结构。如图1(g)所示,UNet ++由不同深度的U-Net组成,其解码器通过重新设计的skip connections以相同的分辨率密集连接。 UNet ++中引入的体系结构具有以下改进优点。首先,UNet ++不容易选择网络深度,因为它在其体系结构中嵌入了不同深度的U-Net。所有这些U-Net都部分共享一个编码器,而它们的解码器则交织在一起。通过在深度监督下训练UNet ++,所有组成的U-Net都将同时接受训练,同时受益于共享的图像表示。这种设计不仅可以提高整体分割性能,而且可以在推理期间剪枝模型。其次,UNet ++不会受到不必要的限制性skip connections的限制,在这种限制就是只能融合来自编码器和解码器的相同比例的特征图。 UNet ++中重新设计了skip connections,那就是在解码器节点处提供了不同比例的特征图,从而使聚合层可以决定如何将skip connections中携带的各种特征图与解码器特征图融合在一起。通过以相同的分辨率密集连接部分U-Net的解码器,可以在UNet ++中实现重新设计的skip connections。我们已经在六个细分数据集和不同深度的多个主干中对UNet ++进行了广泛的评估。我们的结果强有力的表明,通过重新设计的skip connections和深度监督,为UNet ++在语义和实例分割提供了更高的性能。与传统的U-Net架构相比,UNet ++的显着改进归因于重新设计的skip connections和扩展的解码器所提供的优势,这些优势共同使水平和垂直网络上的图像特征逐渐聚合。
总而言之,我们做出了以下五点贡献:
1)我们在UNet ++中引入了一个内置的深度可变的U-Net集合,从而提高了对各种尺寸对象的分割性能,这是对固定深度U-Net的改进(请参阅第II-B节),
2)我们重新设计了UNet ++中的skip connections,从而在解码器中实现了灵活的特征融合,这是对U-Net中仅需要融合相同比例特征图的限制性skip connections的一种改进(请参阅第II-B节)。
3)我们设计了一种方案来剪枝经过训练的UNet ++,在保持其性能的同时加快推理速度(请参阅第IV-C节)。
4)我们发现,同时训练嵌入在UNet ++结构中的多深度U-Net,可以激发在U-Nets结构中协作学习,这与单独训练具有相同体系结构的隔离U-Net相比,可带来更好的性能(请参阅第IV-D节和VC节。
5)我们证明了UNet ++在多个主干编码器上的可扩展性,并进一步适用于包括CT,MRI和电子显微镜在内的各种医学成像模式(请参阅第IV-A节和第IV-B节)。
II. P ROPOSED N ETWORK A RCHITECTURE : UN ET ++
图1显示了UNet ++是如何从原始U-Net演变而来的。 在下文中,我们首先跟踪这种演变,激发对UNet ++的需求,然后解释其技术和实现细节。
A. Motivation behind the new architecture
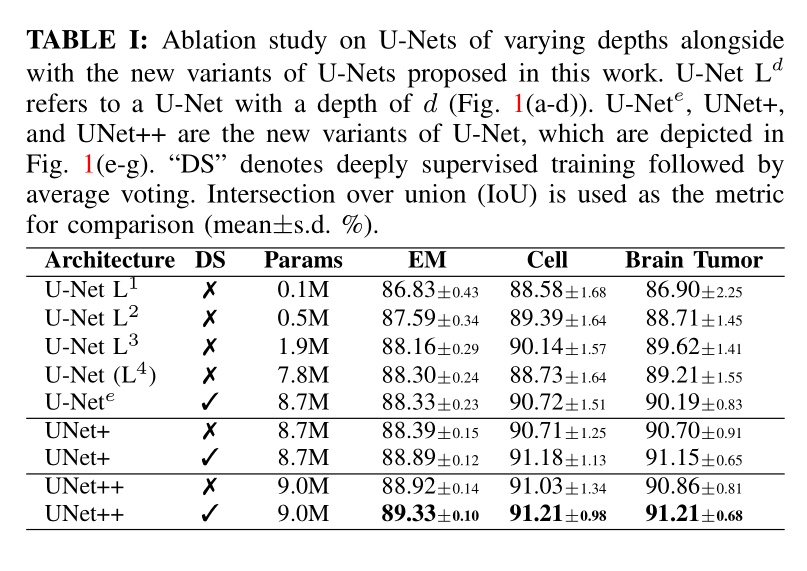
我们已经进行了全面的消融研究,以研究不同深度的U-Nets的性能(图1(a-d))。为了这个目的,我们使用了三个相对较小的数据集,即细胞,EM和脑肿瘤(在III-A节中详细介绍)。表I总结了结果。对于细胞和脑肿瘤的分割,较浅的网络(U-Net L 3)胜过较深的U-Net。另一方面,对于EM数据集,较深的U-Net始终优于较浅的U-Net,但性能提升仅是微不足道的。我们的实验结果提出了两个关键发现:1)更深的U-Net不一定总是更好,2)最佳架构深度取决于手头上数据集的难度和大小。尽管这些发现可能会鼓励进行自动化的神经体系结构搜索,但是这种方法由于有限的计算资源而受到阻碍[19],[20],[21],[22],[23]。另外,我们提出了一个集成架构,该架构将不同深度的U-Net合并为一个统一的结构。我们将此架构称为U-Net e(图1(e))。我们通过为集合中的每个U-Net定义一个单独的损失函数来训练U-Net e,即X 0,j,j∈{1,2,3,4}。我们的深度监控方案不同于深度图像分类和图像分割网络中常用的深度监控;在[24],[25],[26],[27]中,辅助损失函数被添加到沿着解码器网络的节点上,即X 4-j,j,j∈{0,1,2,3,4},而我们将它们应用于X 0,j,j∈{1,2,3,4}。在推理时,对集合中每个U-Net的输出求平均值。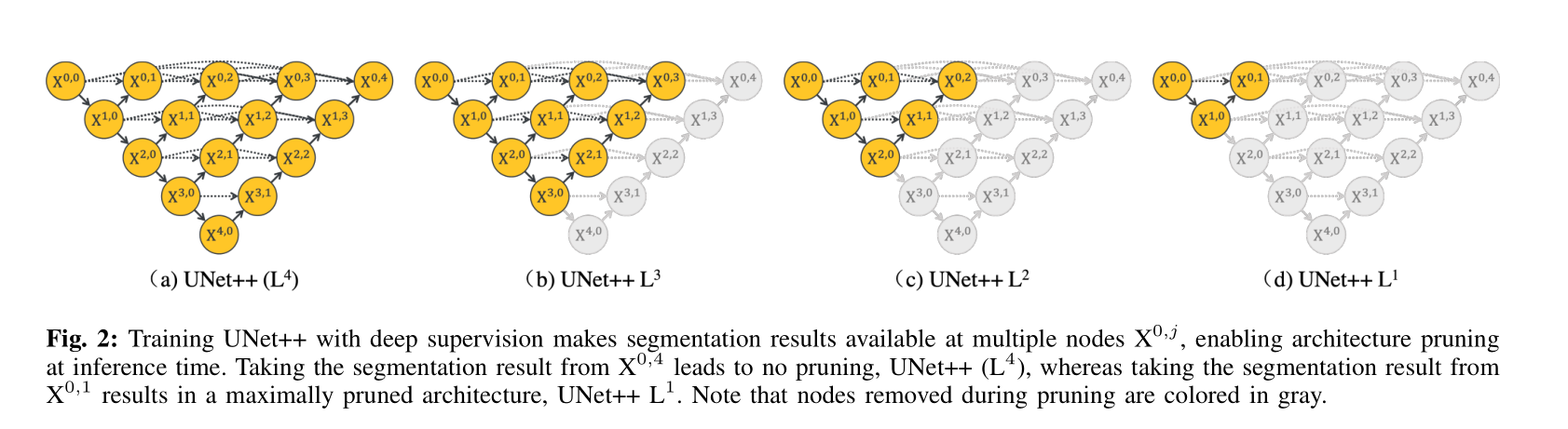
上面概述的集成体系结构(U-Net e)受益于知识共享,因为集成中的所有U-Net尽管他们拥有自己的解码器但也部分共享相同编码器。 然而,该架构仍然遭受两个缺点。 首先,在集成环境下,当解码器已断开连接,较深的U-Net并不会向较浅的U-Net的解码器提供监控信号。 其次,U-Net e中使用的跳过连接的通用设计受到了不必要的限制,要求网络将解码器特征图与仅来自的相同比例的特征图进行组合编码器。 虽然是自然设计,但不能保证相同比例的特征图是特征融合的最佳匹配。
为了克服上述限制,我们从U-Net e中删除了原始的跳跃连接,并连接了集合中的每两个相邻节点,从而形成了一种新的架构,我们称之为UNet +(图1(f))。由于采用了新的连接方案,UNet +连接了不相交的解码器,从而实现了从较深的解码器到较浅的解码器的梯度反向传播。 UNet +展现了在每一个解码器中的每个节点,这些节点都是浅层流中计算的所有特征图的聚合,进一步释放了跳跃连接不必要的限制性行为。尽管在解码器节点上使用聚合的特征图比仅从编码器得到相同比例的特征图的约束要少得多,但仍有改进的空间。我们进一步建议在UNet +中使用密集连接,从而形成最终的架构提案,我们称为UNet ++(图1(g))。通过密集的连接,不仅为解码器中的每个节点提供了最终的聚合特征图,而且还提供了中间的聚合特征图和来自编码器的原始等比例特征图。这样,在解码器节点中的聚合层或许可以学习仅仅去使用相同比例的编码器特征图或者在入口使用收集的所有可用特征图。与U-Net e不同,UNet +和UNet ++对深度监督并不是必须要求的,但是,正如我们稍后将描述的那样,深度监督可以在推理期间剪枝模型,从而导致明显的speedup,而在performance仅适度下降。
B. Technical details
1)网络连通:令x i,j表示节点X i,j的输出,其中i沿编码器索引下采样层,j沿着跳跃连接密集块的卷积层。 由x i,j表示的特征图的堆栈计算如下: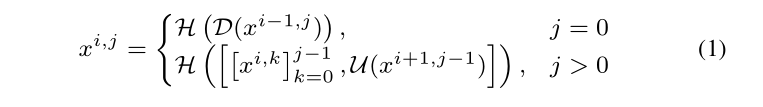
函数H(·)是一个卷积运算,后跟激活函数,D(·)和U(·)表示下采样层和上采样层,[]表示串联层。基本上,如图1(g)所示,级别j = 0的节点仅接收来上一个自编码器层的一个输入;级别j = 1的节点从编码器子网接收两个输入,但在两个连续级别上;并且级别j> 1的节点接收j + 1输入,其中j输入是同一跳跃连接中前j个节点之前的输出,第j + 1输入是来自较低跳跃连接的上采样输出。所有先前的特征图都会累积并到达当前节点的原因,是因为我们沿着每个跳跃连接都使用了密集卷积块。
2)深度监督:我们在UNet ++中引入了深度监督。为此,我们将带有C核的1×1卷积和Sigmoid激活函数附加到节点X 0,1,X 0,2,X 0,3和X 0,4的输出中,其中C是给定数据集中观察到的类的数量。然后,我们定义了一种混合分割损失,包括对于每个语义尺度的像素交叉熵(cross-entropy)损失和 soft dice系数损失。混合损失可以利用两种损失函数必须提供的优势:平滑的梯度和类不平衡的处理[28],[29]。在数学上,混合损失定义为: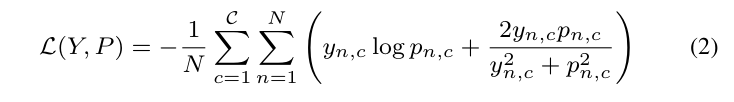
其中 y n,c∈Y 和 p n,c∈P 表示批次中c类和第n个像素的目标标签和预测概率,N表示一个批次中的像素数。然后,对于UNet ++的所有损失函数之后定义为每个独自解码器的混合损失的权重总和:.png),其中d表示解码器的索引。在实验中,我们对每个损失赋予相同的平衡权重ηi,即ηi≡1,并且不针对不同输出监督的真实标签,如高斯模糊。
3)模型剪枝:深度监督可实现模型剪枝。 由于深度监督,UNet ++可以以两种操作模型进行部署:1)集成模型,其中收集所有分割分支的分割结果,然后取其平均值; 2)剪枝模型,其中仅从分割分支之一中选择分割输出 ,其选择决定了模型剪枝的大小和速度增益。 图2显示了如何选择分割分支,以及在不同的剪枝结构中的结果。 具体来说,从X 0,4不会剪枝,然而从X 0,1取得的分割结果则会导致网络的最大剪枝。
III. E XPERIMENTS
A. Datasets
表II总结了本研究中使用的六个生物医学图像分割数据集,涵盖了来自最常用的医学成像模式(包括显微镜检查,计算机断层扫描(CT)和磁共振成像(MRI))的病变/器官。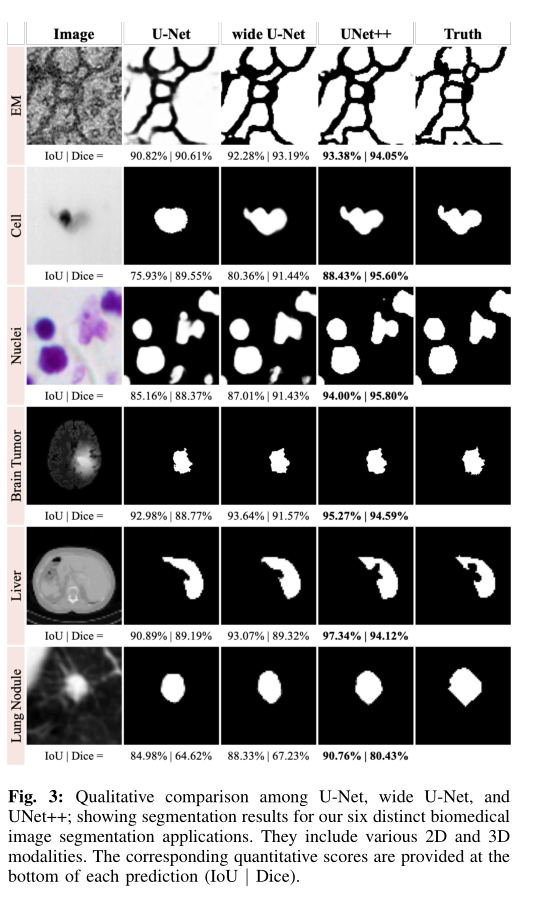
1)电子显微镜(EM):数据集由EM分割挑战[30]作为ISBI 2012的一部分提供。该数据集由果蝇初生幼虫的连续切片透射电子显微镜得到的30张图像(512×512像素)组成 幼虫腹神经索(VNC)。 参考图3中的示例,每个图像都带有对应的完全注释的地面真相分割图,用于细胞(白色)和膜(黑色)。 标记的图像分为训练(24个图像),验证(3个图像)和测试(3个图像)数据集。 训练和推理都是基于96×96 patches进行的,这些patches通过滑动窗口被选择为重叠大小一半。 具体来说,在推论过程中,我们通过在重叠区域进行投票来聚集跨patches的预测。
438/5000
2)细胞:使用Cell-CT成像系统获取数据集[31]。 两名训练有素的专家手动分割了收集的图像,因此数据集中的每个图像都带有两个二进制单元格蒙版。 对于我们的实验,我们选择了354个图像的子集,这些图像在两个专家注释者之间的一致性最高。 然后将选定的图像分为训练(212个图像),验证(70个图像)和测试(72个图像)子集。
3)核:数据集由Data Science Bowl 2018分割挑战赛提供,由670个来自不同模态(明场与荧光)的分割核图像组成。 这是在此工作中使用的带有实例级注释的唯一数据集,其中每个核仁均以不同的颜色标记。 将图像随机分配到训练集(50%),验证集(20%)和测试集(30%)中。 然后,我们使用滑动窗口机制从图像中提取96×96色块,其中32像素的stride用于训练和验证模型,而1像素的stride用于测试。
4)脑瘤:该数据集由BraTS 2013 [32],[34]提供。 为了简化与其他方法的比较,这个模型通过使用来自所有患者的MR扫描图像来训练模型,这些图像是带有Flair(液体衰减反转恢复)的20份高级别(HG)和十份低级别(LG),T1,T1c,和T2。 66,348片 我们通过将切片重新缩放为256×256进一步预处理数据集。 最后,数据集中的30位患者随机分为五张,每张都有六位患者的图像。 然后,我们将这五个折叠随机分配到一个训练集(3个折叠),一个验证集(1个折叠)和一个测试集(1个折叠)中。 真实标签分割具有四个不同的标记:坏死,水肿,非增强性肿瘤和增强性肿瘤。 在BraTS 2013之后,通过将所有四个标签都视为肯定类别,将其标签视为负面类别来完成“完整”评估。
5)肝脏:该数据集由MICCAI 2017 LiTS挑战赛提供,包括331个CT扫描,我们将其分为训练(100例患者),验证(15例患者)和测试(15例患者)子集。ground truth分割提供两个不同的标签:肝脏和病变。 对于我们的实验,我们仅将肝脏视为阳性,将其他肝脏视为阴性。
6)肺结节:该数据集由肺图像数据库协会图像收集(LIDC-IDRI)提供[33],由七个学术中心和八个医学影像公司收集的1018例病例组成。 查明并删除了六起涉及事实真相的案件。 其余案例分为训练(510),验证(100)和测试(408)集。 每种情况都是3D CT扫描,并且结节已被标记为体积二进制掩码。
我们将这个体积重新采样为1-1-1间距,然后在每个结节周围进行了64×64×64的裁剪。 这些3D组用于模型训练和评估。
B. Baselines and implementation
为了进行比较,我们使用原始的U-Net [35]和用于2D分割任务的自定义宽幅U-Net体系结构,以及V-Net [28]和用于3D分割任务的自定义宽幅V-Net体系结构。 我们选择U-Net(或用于3D的V-Net),因为它是图像分割的常见性能基准。 我们还设计了具有与我们类似数量的参数的宽U-Net(或用于3D的宽V-Net)建议的架构。 这是为了确保我们的架构所获得的性能提升不仅仅是因为参数数量的增加。 表III详细列出了U-Net和广泛的U-Net体系结构。 我们进一步比较了UNet ++和UNet +的性能,这是我们的中间体系结构建议。 表III中给出了中间节点中的内核数。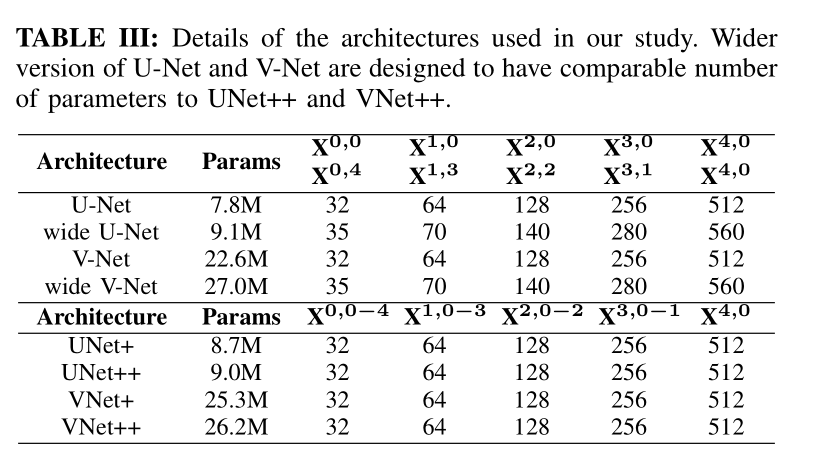
我们的实验是在带有Tensorflow环境下的Keras中实现的。 我们在验证集上使用早期停止机制,以避免过度拟合,并使用Dice系数和Intersection over Union(IoU)评估结果。 可以在附录A中找到替代的度量指标,例如像素灵敏度,特异性,F1和F2分数以及统计分析。Adam被用作优化器,学习率为3e-4。 UNet +和UNet ++都是从原始U-Net体系结构构建的。 所有实验均使用三个NVIDIA TITAN X(Pascal)GPU(每个GPU具有12 GB内存)执行。
IV. R ESULTS
A. Semantic segmentation results
表IV比较了U-Net,宽U-Net,UNet +和UNet ++的数量参数和分割结果对基于六个分割任务的研究我们采用了IoU( (mean±s.d))来衡量。如图所示,宽的U-Net始终优于U-Net。这种改进归因于宽U-Net中的大量参数。在所有六个任务,没有深层监督的UNet ++在U-Net和宽U-Net上均实现了显着的IoU增益,如如在神经元结构(↑0.62±0.10,↑0.55±0.01),(↑0.62±0.10,↑0.55±0.01),细胞(↑2.30±0.30,↑2.12±0.09),没有深层监督的UNet ++在U-Net和宽U-Net上均获得了显着的IoU增益),细胞核(↑1.87±0.06,↑1.71±0.06),脑瘤(↑2.00±0.87,↑1.86±0.81),肝脏(↑2.62±0.09,↑2.26±0.02)和肺结节(↑5.06±1.42) ,↑3.12±0.88)分割。使用深度监督和平均投票可以更进一步改善UNet ++,将IoU提升多达0.8点。特别是,神经元结构和肺结节分割在深度监督中受益最大,因为它们在EM和CT切片中以不同的比例出现。但是,深度监督最多只能对其他数据集有效。图3描述了U-Net,宽U-Net和UNet ++的结果之间的定性比较。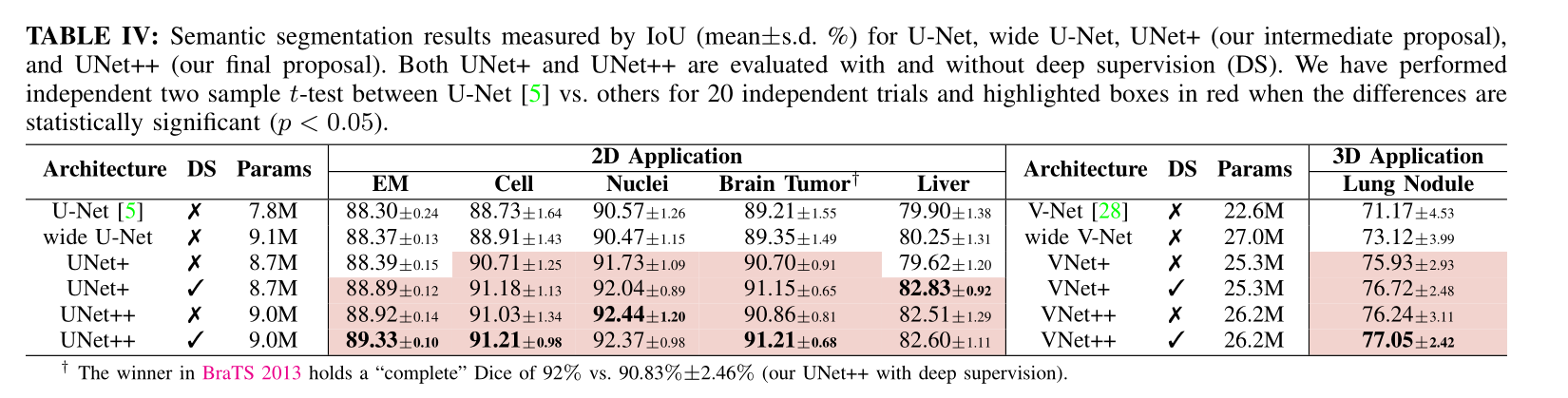
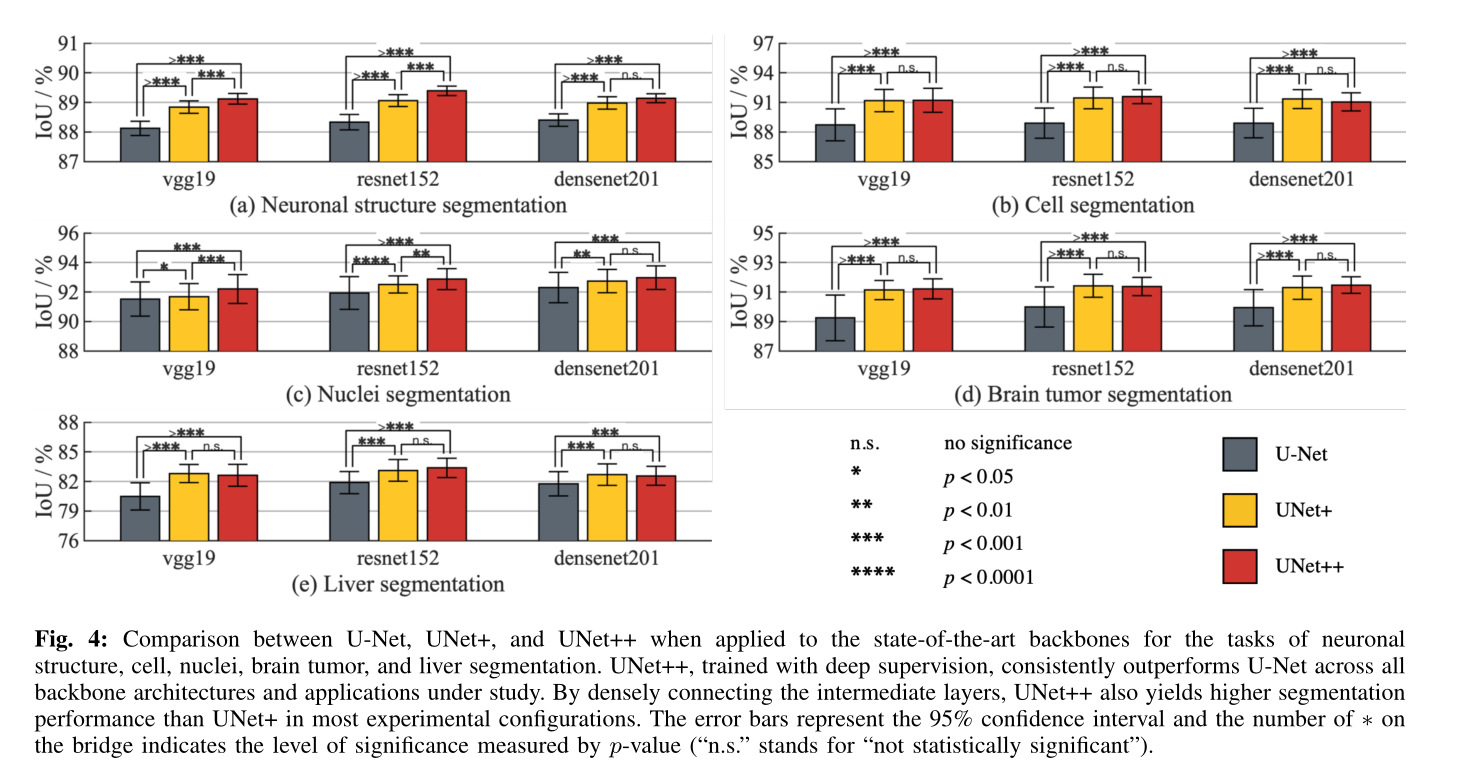
我们更进一步研究了UNet++在语义分割中的扩展性,通过将重新设计的跳跃连接应用于现代CNN架构数组中:vgg-19 [36],resnet-152 [8]和densitynet-201 [9],。具体来说,我们通过添加解码器子网将上述每个体系结构转变为U-Net模型,然后用重新设计UNet++的连接替换了U-Net的普通跳过连接。为了进行比较,我们还使用上述骨干架构训练了U-Net和UNet +。为了进行全面比较,我们使用了EM,细胞,细胞核,脑肿瘤和肝脏分割数据集。如图4所示,在所研究的所有主干架构和应用程序中,UNet ++始终优于U-Net和UNet +。通过20个试验,我们进一步基于U-Net,UNet +和UNet ++中每对的独立两样本t检验提供了统计分析。我们的结果表明,UNet ++是对U-Net的有效,与主干无关的扩展。为了促进可重复性和模型重用,我们针对各种传统和现代骨干架构发布了U-Net,UNet +和UNet ++的实现1。
B. Instance segmentation results
实例分割包括对所有对象实例进行分割和区分。 因此,比语义分割更具挑战性。 我们使用Mask R-CNN [12]作为实例分割的基线模型。 Mask R-CNN以特征金字塔网络(FPN)为骨干,去生成一个多尺度对象提案,然后通过专用的分割分支为收集的提案输出分割Mask。 我们修改了Mask R-CNN,通过用重新设计的UNet ++跳跃连接来取代FPN的普通跳过连接。 我们将此模型称为Mask RCNN ++。 我们在我们的实验中,使用resnet101作为Mask R-CNN的主干。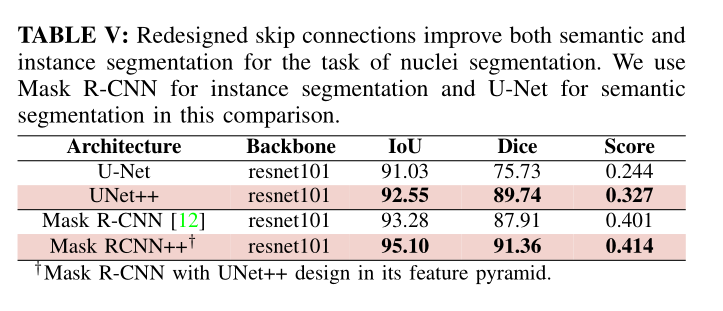
表V比较了Mask R-CNN和Mask RCNN ++在核分割方面的性能。我们之所以选择Nuclei数据集,是因为图像中可以存在多个核仁实例,在这种情况下,每个实例都以不同的颜色进行注释,因此被标记为不同的对象。因此,该数据集既适合于将所有核实例都视为前景类的语义分割,也适合于将每个单独的核分别去分割的实例分割。如表V所示,Mask RCNN ++的表现优于其同类产品,IoU提高了1.82分(从93.28%提高到95.10%),Dice系数提高了3.45分(从87.91%提高到91.36%),排行榜得分提高了0.013分(0.401到0.414)。为了更好地了解这种性能,我们还训练了一个U-Net和UNet ++模型,用于使用resnet101主干进行语义分割。如表V所示,Mask R-CNN模型比语义分割模型具有更高的分割性能。此外,正如预期的那样,UNet ++在语义分割方面优于U-Net。
C. Model pruning
一旦对UNet ++进行了训练,则在推理时深度d的解码器路径就与深度d + 1的解码器路径完全独立。因此,我们可以完全删除深度d + 1的解码器,从而获得训练后的较浅版本。由于引入了深度监督,因此深度为d的UNet ++。这种剪枝可以显着减少推理时间,但是分割性能可能会下降。因此,剪枝级别应通过在验证集中评估模型的性能来确定。我们在图5中对UNet ++的IoU推理速度进行了权衡研究。我们使用UNet ++ Ld表示在深度d处修剪的UNet ++(有关更多详细信息,请参见图2)。可以看出,UNet ++ L3的平均推理时间减少了32.2%,内存占用减少了75.6%,而IoU仅降低了0.6点。更积极的修剪进一步减少了推理时间,但代价是IoU明显下降。更重要的是,由于现有的深度卷积神经网络模型的存在,这种观察有可能对移动设备上的计算机辅助诊断(CAD)产生重要影响。计算量大且占用大量内存。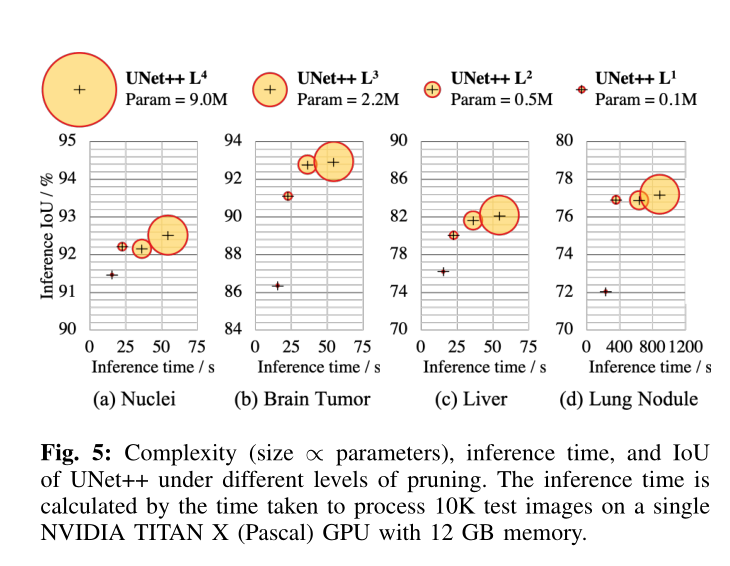
D. Embedded vs. isolated training of pruned models
从理论上讲,可以通过两种方式训练UNet ++ Ld:1)嵌入式训练,其中训练完整的UNet ++模型,然后在深度d上剪枝以得到UNet ++ L d,2)孤立训练,其中UNet ++ L d在没有任何深度编码和解码器的节点上交互而单独训练。 参见图2,在深度监督的情况下子网的嵌入式训练包括训练所有图节点(黄色和灰色),但是在推理期间,我们仅使用黄色子网。 相反,孤立的训练包括从图形中删除灰色节点,仅基于黄色子网络进行训练和测试。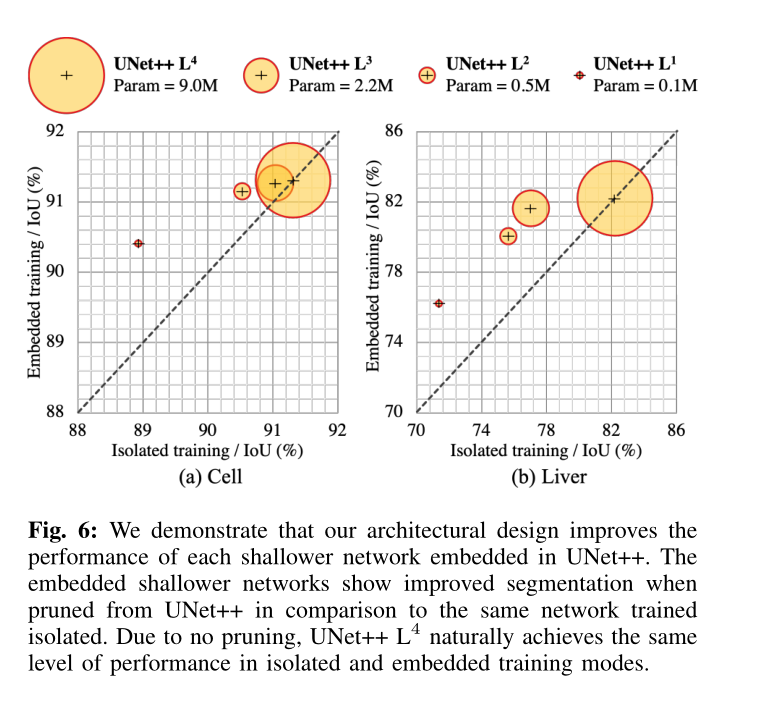
我们比较了图6中两个数据集上在孤立训练和嵌入式训练这两个方案中,不同级别的UNet ++剪枝。我们发现,与孤立训练相同的结构相比,UNet ++ L d的嵌入式训练产生了更高的性能模型。 当将完整的UNet ++剪枝为UNet ++ L 1时,在主动剪枝下观察到的优势更为明显。 特别是针对肝脏分割的UNet ++ L 1嵌入式培训与孤立的培训计划相比,IoU达到5分增加。 这一发现表明,来自下游深处的监督信号可以训练性能更好的浅层模型。 这一发现还与知识提炼有关,在知识提炼中,由较深的教师网络学习的知识由较浅的学生网络学习。
V. D ISCUSSIONS
A. Performance analysis on stratified lesion sizes
图7比较了U-Net和UNet ++用于分割不同大小的脑肿瘤。 为避免图中混乱,我们将肿瘤按大小分为七个桶。 如图所示,UNet ++在所有存储桶中始终优于U-Net。 我们还基于20个不同的试验对每个存储桶进行t检验,以衡量改进的重要性,结论是7个比较中有5个具有统计学意义(p <0.05)。 UNet ++分割不同大小的肿瘤的能力归因于其内置的U-Net集成,它可以基于多感受野网络进行图像分割。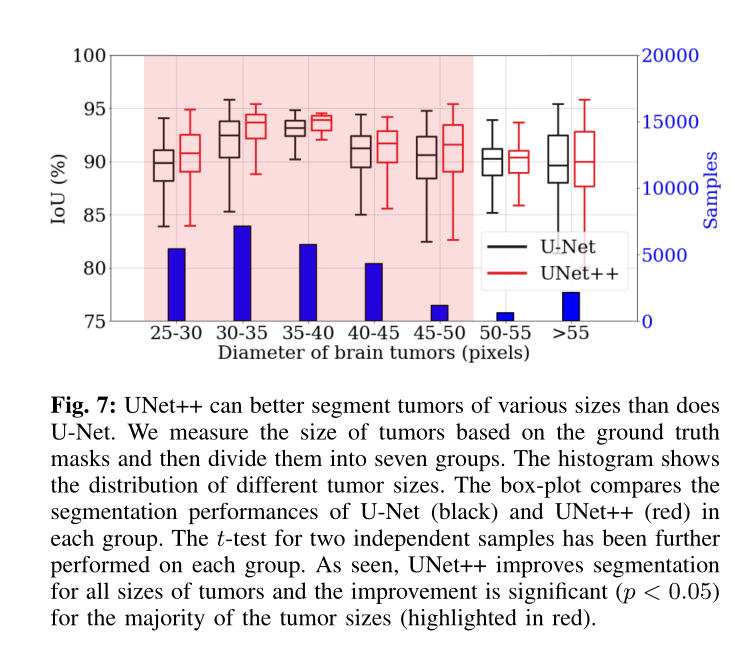
B. Feature maps visualization
在第II-A节中,我们解释了重新设计的跳跃连接可以将语义丰富的解码器特征图与来自体系结构中间层的语义级别不同的特征图进行融合。 在本节中,我们通过可视化中间特征图来说明重新设计的跳跃连接的优势。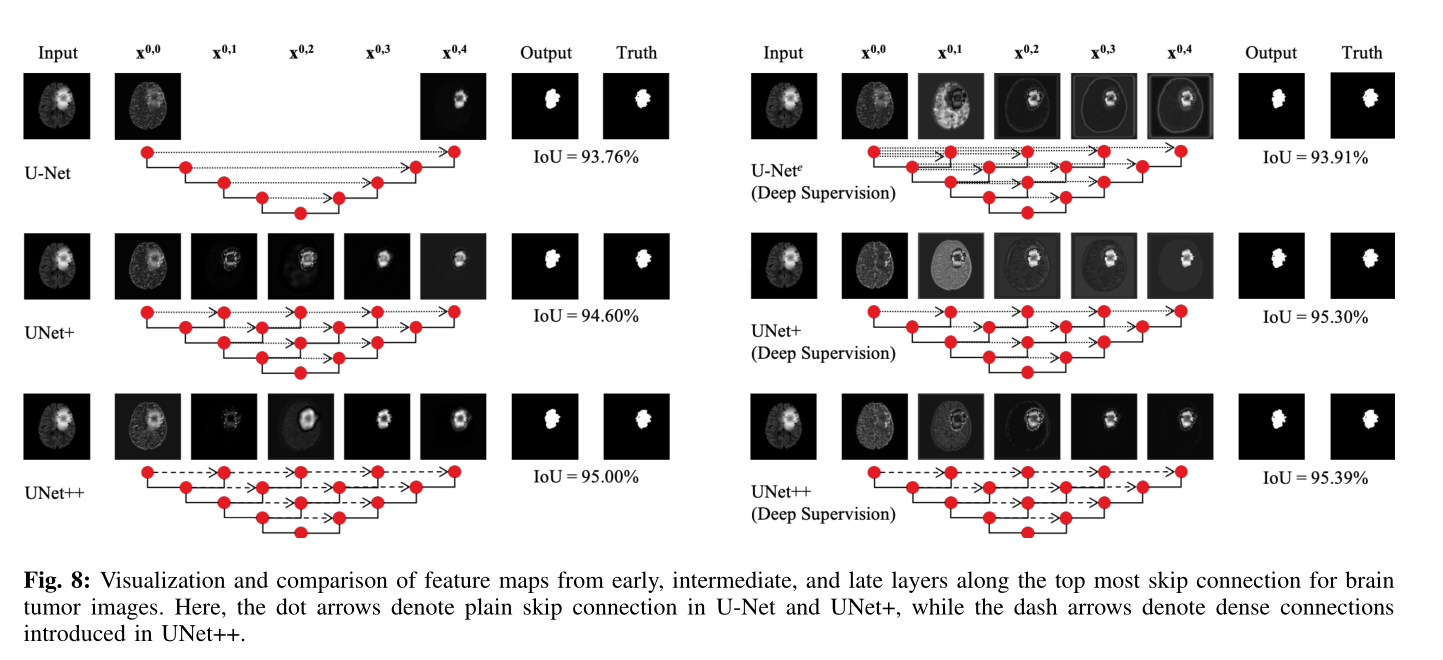
图8示出了沿着脑肿瘤图像的最顶部跳过连接(即,X 0,i)的来自早期,中间和晚期层的代表性特征图。对于某一层的代表特征图是通过平均所有特征图获得的。还应注意,仅使用附加深度解码器层(X 0,4)的损失函数来训练图8左侧的体系结构,而图8右侧的体系结构是在深度监督下进行的。请注意,这些功能图不是最终的输出。我们在每个解码器分支的顶部附加了一个额外的1×1卷积层,以形成最终的分割。我们发现,U-Net中间层的输出在语义上是不同的而对于UNet +和UNet ++,输出是逐渐形成的。 U-Net中的节点X 0,0的输出经过轻微的变换(仅很少的卷积运算),而X 1,3的输出(X 0,4的输入)几乎经历了每个变换(四个下采样和三个上采样)。因此,X 0,0和X 1,3的表示能力之间存在很大的差距。因此,简单地将X 0,4和X 1,3的输出串联起来并不是最佳解决方案。相反,在UNet +和UNet ++中重新设计的跳跃连接有助于逐步完善分段结果。我们在附录B部分中进一步介绍了所有六个医疗应用程序的学习曲线,揭示了在UNet ++中添加密集连接鼓励了更好的优化并降低了验证损失。
C. Collaborative learning in UNet++
协作学习称为在同一训练数据上同时训练同一网络的多个分类器。人们发现它可以提高深度神经网络的泛化能力[37]。 UNet ++通常体现协作学习是通过聚合多深度网络并监督每个组成网络的分割。此外,分割头(例如,图2中的X 0,2)从强(来自真实标签的损失)和软(从邻近的较深节点传播的损失)监管接收梯度。结果,较浅的网络改善了其分割(图6)并提供了向更深入的同行提供更多信息。基本上,更深层的网络和浅层网络通过UNet ++中的协作学习使彼此正规化。与单独训练作为隔离网络的单独训练相比,一起训练多深度的嵌入式网络可以提高分割效果,这在IV-D节中很明显。 UNet ++的嵌入式设计使其可用于辅助培训,多任务学习和知识提炼[17],[38],[37]。
VI. R ELATED W ORKS
下面,我们回顾与重新设计的跳跃连接,特征聚合和深度监督有关的工作,这些是我们新体系结构的主要组成部分。
A. Skip connections
跳跃连接最早是在Long等人的开创性工作中引入的。 [39]他们提出了用于语义分割的全卷积网络(FCN)。不久之后,Ronneberger等人建立了跳跃连接。 [35]提出了用于医学图像语义分割的U-Net架构。但是,FCN和U-Net架构在上采样解码器特征图的处理方式上有所不同与来自编码器网络的相同比例的特征图相融合。当FCN [39]使用求和运算进行特征融合时,U-Net [35]将特征串联起来,然后再应用卷积和非线性。跳跃连接已显示出有助于恢复完整的空间分辨率的能力,从而使完全卷积的方法适用于语义分割[40],[41],[42],[43]。跳跃连接已进一步用于现代神经体系结构中,例如残差网络[8],[44]和密集网络[9],梯度流和改善分类网络的整体性能。
B. Feature aggregation
聚合分层特征的探索最近已经成为研究的主题。 Fourure等。 [45]提出了GridNet,它是一种编码器-解码器体系结构,其中特征图以网格方式进行布线,概括了几种经典的分割体系结构。尽管GridNet包含多个具有不同分辨率的流,但是它缺少跳跃连接之间的上采样层。因此,它不代表UNet ++。全分辨率残差网络(FRRN)[46]采用两流框架,其中全分辨率信息承载在一个流中,上下文信息承载在另一个池流中。在[47]中,提出了两种改进的FRRN版本,即具有28.6M参数的增量MRRN和具有25.5M参数的密集MRRN。但是,这些2D架构的参数数量与我们的3D VNet ++相似,并且参数是2D UNet ++的三倍;因此,仅将这些体系结构升级为3D方式可能不适用于常见的3D体积医学成像应用。我们想指出的是,我们重新设计的密集跳跃连接与MRRN中使用的连接是完全不同的,MRRN由一个公共的剩余流组成。另外,将MRRN的设计应用于其他骨干编码器和元框架(例如Mask R-CNN [12])也不灵活。 DLA 2 [48],在拓扑上等效于我们的中间体系结构UNet +(图1(f)),按顺序连接相同分辨率的特征图,而无需使用U-Net中的长时间跳过连接。我们的实验结果表明,通过紧密地连接各层,UNet ++可以实现比UNet + / DLA更高的分段性能(请参见表IV)。
C. Deep supervision
他等。 [8]建议网络深度d可以充当正则化器。 Lee等。文献[27]证明了深度监督层可以提高隐藏层的学习能力,并强制中间层学习判别特征,从而实现网络的快速收敛和规范化[26]。 DenseNet [9]以隐式方式执行类似的深度监控。深度监督也可以在类似U-Net的体系结构中使用。 Dou等。 [49]通过结合来自特征图的不同分辨率的预测来引入深度监督,这表明它可以克服潜在的优化难题,从而达到更快的收敛速度和更强大的判别能力。朱等。 [50]在他们提出的体系结构中使用了八个额外的深度监督层。但是,我们的嵌套网络更适合在深度监督下进行训练:1)多个解码器自动生成全分辨率分割图; 2)网络被嵌入到各种不同深度的U-Net中,从而掌握了多分辨率特征; 3)紧密相连的特征图有助于平滑梯度流并提供相对一致的预测蒙版; 4)高维特征会通过反向传播对每个输出产生影响,使我们能够在推理阶段修剪网络。
D. Our previous work
我们首先在DLMIA 2018论文[51]中介绍了UNet ++。此后,UNet ++被研究界迅速采用,可以作为比较的可靠基准[52],[53],[54],[55],也可以作为比较的基础。开发新语义分割体系结构的灵感来源[56],[57],[58],[59],[60],[61];它也已用于多种应用,例如在生物医学图像[62],[63],自然图像[64]和卫星图像[65],[66]中分割对象。最近,Shenoy [67]针对“接触预测模型PconsC4”的任务独立而系统地研究了UNet ++,证明了在广泛使用的U-Net上的重大改进。尽管如此,为了进一步增强我们自己的UNet ++,当前的工作对我们以前的工作进行了一些扩展:(1)我们对网络深度进行了全面的研究,从而激发了对所提议的体系结构的需求(第II-A节); (2)我们将修剪的UNet ++各个级别的嵌入式训练方案与孤立的训练方案进行了比较,发现与单独进行单独训练相比,训练多深度的嵌入式U-Net可以提高性能(第IV-D节); (3)通过增加用于脑肿瘤分割的新的磁共振成像(MRI)数据集来加强我们的实验(第四节); (4)我们证明了UNet ++在Mask R-CNN中的有效性,从而产生了一个更强大的模型,即Mask RCNN ++(第IV-B节); (5)我们研究了UNet ++对多个高级编码器主干进行语义分割的可扩展性(第IV-A节); (6)我们研究了UNet ++在分割大小不同的病变方面的有效性(第V-A节);和(7)可视化沿退位跳过的特征传播连接以说明性能(第V-B节)。
VII. C ONCLUSION
我们提出了一种名为UNet ++的新颖体系结构,用于更精确的图像分割。 我们的UNet ++改进的性能归因于其嵌套结构和重新设计的跳过连接,旨在解决U-Net的两个关键挑战:1)最佳架构的深度未知; 2)跳跃连接的不必要的限制性设计。 我们已经使用六个不同的生物医学成像应用程序对UNet ++进行了评估,并证明了针对语义分割和元框架(例如实例分割)的各种最新骨干网的性能持续改进。

